AVALIAÇÃO TEÓRICA DA AFINIDADE RELATIVA DE INIBIDORES DE TROMBINA VIA PERTURBAÇÃO DA ENERGIA LIVRE
Cristiano Ruch Werneck Guimarães* (PG) & Ricardo Bicca de Alencastro (PQ)
Laboratório
de Modelagem Molecular, Departamento de Química
Orgânica,
Instituto de Química, Universidade Federal
do Rio de Janeiro, Cidade Universitária, Bloco A, lab. 609,
Rio de Janeiro, RJ 21949-900
e-mail:cris@pc142.iq.ufrj.br
palavras-chave: inibidores de trombina, dinâmica molecular, perturbação da energia livre
A trombina é uma serinil-protease que desempenha papel central na coagulação sangüínea. Esta enzima é responsável pela conversão do fibrinogênio em fibrina, sendo também o estimulante mais potente da agregação plaquetária. Desta forma, sua inibição tornou-se o alvo principal no desenvolvimento de novos agentes antitrombóticos. Dentre os inibidores do centro ativo, podemos citar a benzamidina1 e a 4-(3-carbóxi-2-oxo-1-propil)-benzamidina.1 No presente trabalho, apresentamos resultados parciais da avaliação da diferença de energia livre de complexação à trombina destes inibidores e entre estes e novos candidatos. Para isto, adotamos a estratégia do cálculo de energia livre relativa entre dois estados, empregando o algoritmo FDTI2 (Discover 2.9.7-InsightII-MSI). A diferença de energia livre da transformação entre dois estados (DAtransf.) pode ser descrita pela equação:
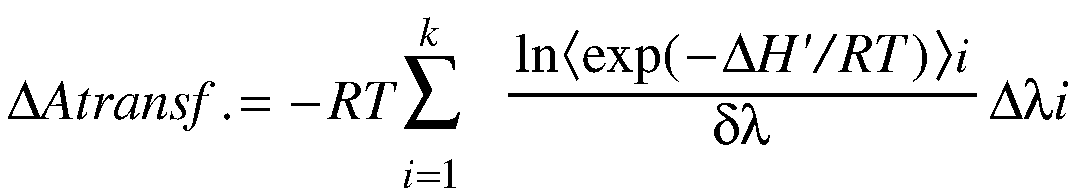
em
que R é a constante dos gases, T é a
temperatura absoluta, DH’
= Hli+dl
– Hli
,
<
>i
refere-se à média em relação a um
ensemble N,V,T do estado representado pelo Hamiltoniano Hli
, l
é o
parâmetro de acoplamento, k é o número
de quadraturas e Dli
é um valor que depende do esquema de integração
numérica usado. O parâmetro de acoplamento l
introduzido no método FDTI quebra o cálculo de energia
livre em janelas, cada qual envolvendo um intervalo suficientemente
pequeno em l para
propiciar um bom recobrimento no espaço de fase e, assim,
permitir que a energia livre seja calculada acuradamente. Este
parâmetro pode variar de 0 (100% do estado inicial) a 1 (100%
do estado final). Em alguns casos,
quando a diferença estrutural entre os dois estados é
significativa, além da introdução do parâmetro
de acoplamento l
para cada transformação, é necessário o
uso de estados intermediários que permitam uma transformação
estrutural mais suave. Deste modo,
a estratégia descrita previamente permite a construção
do ciclo termodinâmico abaixo, que viabiliza o cálculo
de diferença de energia livre de complexação à
trombina entre os dois inibidores citados acima. No ciclo
termodinâmico, podemos verificar que só são
necessárias as quantidades DAtransf.i(comp.)
e DAtransf.i(sol.),
transformação i no complexo solvatado e em
solução, para o cálculo da diferença de energia livre de complexação (DDAcomp.) entre os inibidores.
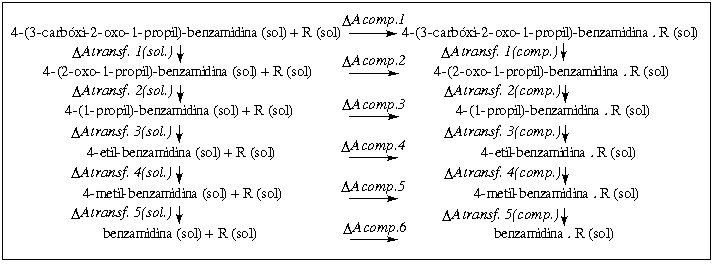
As
transformações foram realizadas usando técnicas
de dinâmica molecular em condições N,V,T
(T = 300 K), aplicando o modelo de solvatação
esférico
(r = 16 Å). Empregamos um potencial
semi-harmônico (k = 0,5 kcal/mol Å2
para
r > 16 Å) para prevenir a "evaporação"
do solvente, um corte esférico de 11 Å para as
interações não ligadas e o campo de força
CVFF.3 Os resultados de DDAcomp
encontram-se dentro da ordem de grandeza esperada para as
transformações 2, 3, 4 e 5, o que não acontece
para a transformação 1 (Tabela). Isto provavelmente é
resultado da solvatação inadequada da carga positiva
gerada na transformação 1. Assim, para
permitir a comparação dos inibidores propostos no
presente trabalho com o inibidor
4-(3-carbóxi-2-oxo-1-propil)-benzamidina, a transformação
1 será repetida com um modelo de solvatação
esférico de maior raio. Apesar disto, com os resultados das
transformações 2, 3, 4 e 5 podemos verificar que com
exceção do inibidor 4-metil-benzamidina, todos os novos
candidatos são melhores que a benzamidina.
|
Transformação i ® |
1 |
2 |
3 |
4 |
5 |
|
DAtransf.i(sol.) |
29,01 |
42,84 |
-15,09 |
-28,90 |
-9,84 |
|
DAtransf.i(comp.) |
58,27 |
44,32 |
-15,34 |
-27,04 |
-11,16 |
|
DDAcomp. |
29,26 |
1,48 |
-0,20 |
1,98 |
-1,32 |
1 - Babine, R. E. & Bender, S. L., 1997 Chem. Rev., 97 (5), 1359.
2 - Mezei, M. et al., 1986 Ann. N. Y. Acad. Sci., 482, 1.
3 - Dauber-Osguthorpe, P. et al., 1988 Proteins: Struct. Funct. Gen., 4, 31.
(FAPERJ, CNPq)