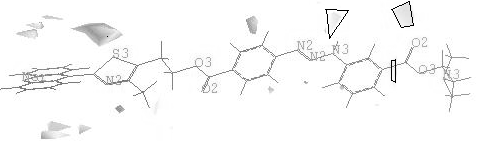
Figura 1: Mapa de contorno energético para as sondas água e "dry". Observam-se interações a 6,5 Kcal/mol para a sonda água (contornada por polígonos) e a 2,0 Kcal/mol para a sonda "dry".
Rogério de Oliveira (IC), Carlos Alberto Montanari (PQ)
Núcleo de Estudos em Química Medicinal,
Departamento de Química,
Instituto de Ciências Exatas, Universidade
Federal de Minas Gerais
palavras-chave: GRID, B-DNA, combilexinas
A necessidade cada vez maior de se obter moléculas anti-cancerígenas mais eficientes é facilmente demonstrada pela quantidade de informações que se pode obter sobre o assunto, além do agregamento dos dados das mais variadas fontes de pesquisa ser cada vez maior. A procura de moléculas com esta ação biológica está centrada basicamente em moléculas como o berenil (análogo da pentamidina), e também a substituição por grupos químicos apropriados como a acridina. Sabe-se hoje por estudos biofísicos que a interação destas moléculas dá-se na fenda menor do B-DNA, onde os análogos estudados apresentam-se ligados preferencialmente às regiões AT do B-DNA1.
As moléculas que contém a acridina ligada aos análogos do berenil já se apresentam complexados na fenda menor do B-DNA com a acridina tendo um papel intercalante importante para a atividade biológica das moléculas, além da diferenciação da potência devida ao resíduo dos análogos. Ambos os derivados têm como característica básica a interrupção da replicação do DNA. Nesta fase do projeto, um novo campo de força está sendo empregado. Trata-se do campo de força GRID2,3.
Este trabalho tem como objetivo a geração dos campos moleculares lipofíllicos, usando as sondas químicas água (hidrofílica) e "dry" (hidrofóbica) e também a racionalização do efeito hidrofóbico.
A estratégia do uso do campo de força GRID foi estabelecida tendo-se em vista que esse procedimento é capaz de descrever as energias de interação entre certos grupos químicos (sondas) e as moléculas em estudo.
O algoritmo de análise do programa estabelece uma grade tridimensional ao redor e através da molécula definida como alvo e para cada ponto da grade uma dentre as várias sondas escolhida é posicionada. A partir deste posicionamento e de posse das distâncias entre os átomos da sonda e os do alvo, o programa calcula a energia potencial Exyz. É feito então novo posicionamento da sonda em outro ponto da grade e novo valor de energia potencial é calculado. O programa procede sucessivamente desta maneira até o esgotamento de todos os pontos da grade tridimensional.
As combilexinas foram geradas através do programa MacroModel, na primeira fase deste projeto, e então foram levadas para o programa Great, que é um gerenciador dos programas Grin e GRID. O programa Great lê as estruturas e faz algumas correções que por ventura se tornem necessárias. Depois de se executar o Grin, executa-se o GRID, utilizando como arquivos de entrada aqueles que são saída do Grin.
Selecionaram-se as sondas químicas para o estudo de suas interações. As sondas utilizadas foram: água e "dry". Depois de executado o programa GRID, converteram-se os dados fornecidos em arquivos gráficos que podem ser visualizados através do programa Sybyl.
Como resultados preliminares temos as energias de
interação entre as sondas e as moléculas em estudo
e também a geração dos mapas de contornos energéticos.
A figura 1 mostra um mapa energético da interação
das sondas água e "dry" (lipofílica) com uma das combilexinas
estudadas, em sua conformação estendida (tipo Raios-X, gerada
pelo programa Corina), para que se tenha uma melhor visualização.
Observam-se interações tanto na região da acridina
assim como na região análoga ao berenil.

Conclui-se que as combilexinas em estudo podem interagir com o B-DNA reversivelmente, uma vez que as interações lipofílicas são relativamente fracas. Nota-se também a robustez do programa GRID e do método de novo no planejamento racional de fármacos.
Referências Bibliográficas: